PythonでMRI/CT/PETなどの医用画像を扱うことができるNiBabelの導入方法と使い方

こんにちは、個人事業OguLinks代表の小倉(@jun_ogulinks)です。
医用画像を普段扱っている方で、画像処理をPythonで自動化したいと考える人は多いと思います。
Pythonには、MRI/CT/PETといった医用画像を扱うためのパッケージとしてNiBabelがあります。
この記事ではNiBabelの導入方法と、その簡単な使い方について紹介します。
この記事を読むことで以下のことが分かるようになっています。
- NiBabelとは何か、その概略
- AnacondaへのNiBabelのインストール方法
- NiBabelでのNIfTIファイルの読み込みと書き出し(保存)の方法
NiBabelとは何か?

まずNiBabelの公式サイトは こちら になります。
公式サイトには以下のように紹介されています。
This package provides read +/- write access to some common medical and neuroimaging file formats, including: ANALYZE (plain, SPM99, SPM2 and later), GIFTI, NIfTI1, NIfTI2, CIFTI-2, MINC1, MINC2, AFNI BRIK/HEAD, MGH and ECAT as well as Philips PAR/REC. We can read and write FreeSurfer geometry, annotation and morphometry files. There is some very limited support for DICOM. NiBabel is the successor of PyNIfTI.
The various image format classes give full or selective access to header (meta) information and access to the image data is made available via NumPy arrays.
これを要約すると以下のようになります(DeepL翻訳まま)。
- 一般的な医療およびニューロイメージングファイル 形式への読み込み/書き込みアクセスを提供
- FreeSurferのジオメトリ、アノテーション、モルフォメトリファイルの読み込みと書き込みが可能
- ヘッダー(メタ)情報へのアクセスを提供
- 画像データへのアクセスはNumPyの配列を介して利用可能
NiBabelで読み込んだ画像ファイルはNiBabel専用のフォーマットとして取り扱うことになります。
NiBabel専用フォーマットでできることもありますが、NiBabelの組み込みメソッドを使わずに独自の処理をしたい場合にはNumPyの配列に変換して使うことになります。

画像をNumPyの配列として扱うことができるというのは非常にありがたいですね。
また、対応ファイルも非常に多いです。
- ANALYZE (plain, SPM99, SPM2 and later)
- GIFTI
- NIfTI1, NIfTI2
- CIFTI-2
- MINC1, MINC2
- AFNI BRIK/HEAD
- MGH and ECAT
- Philips PAR/REC など
DICOMについてはごく限られた範囲でのみ対応しているということなので、あまり使わない方が良いでしょう。
私は普段、MRI/CT/PETのDICOMを4DのNIfTIに変換して使っています。
変換方法は別の記事で詳しく紹介したいと思いますので、ここでは変換に使っているソフトウェアの名前だけに留めておきます。
変換にはdcm2niixというMRIcroGLに含まれる機能を使っています。
MRIcroGLを起動後、メニューバーの Import > Convert DICOM to NIfTI の順でクリックするとdcm2niixを起動することができます。
以降はこの変換したNIfTI形式の画像ファイルを例に説明していきます。
NiBabelのインストール方法

インストール方法はいくつかあります。
公式サイトでは pip を使ったインストール方法が紹介されています。
pip を使ってインストールしたい方は公式サイトを参照してください。
この記事ではAnacondaを使ってインストールする方法を紹介します。
まず、前提として知っておかなければならないことがあります。
それは NiBabel パッケージはPythonの標準のリポジトリには登録されていないため、いきなりインストールはできないということです。
こちらのAnaconda.orgのページを見ていただくと分かりますが、NiBabelはconda-forgeに登録されているパッケージになります。
そのため、まずは自身のAnacondaにconda-forgeを設定しておいてください。
設定の仕方はこちらの記事で紹介しています。

conda-forgeの設定が完了したら、NiBabelのインストールに移ります。
一般的なパッケージのインストール方法と同じになります。
下図のようにパッケージ表示リストを All に切り替えてから検索し、チェックを入れたらApplyボタンをクリックすればOKです。
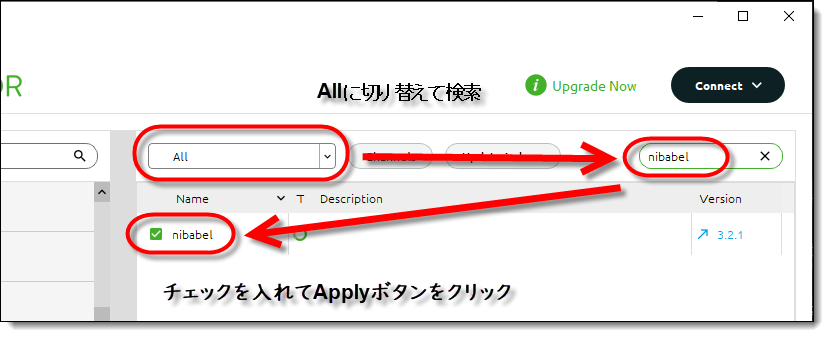
もし、一般的なパッケージのインストール方法が分からないということであれば、こちらの記事を参照ください。
これでAnaconda上Python仮想環境への、NiBabelのインストールは完了です。
Google ColaboratoryでNiBabelを使うためには
セキュリティや倫理的な観点からローカルで作業する方が多いかと思いますが、中にはオンライン上でサクッと処理したいんだ!という方もいるかと思います。
ということで、Google Colaboratory(以下、Google Colab)でのNiBabelの導入方法についてもお話しします。
Google ColabでNiBabelを導入するために必要な作業は、
ありません。
実はGoogle Colabの場合、何もしなくてもNiBabelが使えます。
まさかこんな専門的なパッケージを読み込むだけで使えるとは思っていませんでした。
毎回システムコマンドを使ってインストールする必要がないのはありがたいですね。
では次に実際の使い方について説明します。
NiBabelでのNIfTIファイルの読み込み方法

実際にNiBabelでNIfTIファイルの読み込みを行ってみます。
今回使うNIfTIファイルは例のごとくSmall Animal Molecular Imaging Toolbox (SAMIT)に含まれているラットのMRI T2Wとします(ファイル名:Schwarz_T2w.nii)。
このラットMRI T2W画像ファイルについてはこちらの記事内で文献リンクを付けていますので興味があれば見てみてください。
ではまず、パッケージの読み込みを行います。
NiBabelのほかにnumpyも読み込んでおきます。
import numpy as np
import nibabel as nibここから先は、カレントディレクトリに Schwarz_T2w.nii がある前提で進めていきます。
画像の読み込みは NiBabel の loadメソッドを使います。
以下のようにファイル名を引数に与えるだけです。
# load NIfTI image
niimg = nib.load("Schwarz_T2w.nii")niimgを実行すると nibabel.Nifti1Image という型であることが分かります。
これがNiBabel専用フォーマットになります。

print(niimg) を実行することで以下のヘッダー情報を表示できます。
<class 'nibabel.nifti1.Nifti1Image'>
data shape (96, 120, 96)
affine:
[[ 0.19999999 0. 0. -9.39999962]
[ 0. 0.19999999 0. -16.79999924]
[ 0. 0. 0.19999999 -13.99999905]
[ 0. 0. 0. 1. ]]
metadata:
<class 'nibabel.nifti1.Nifti1Header'> object, endian='<'
sizeof_hdr : 348
data_type : b''
db_name : b''
extents : 0
session_error : 0
regular : b'r'
dim_info : 0
dim : [ 3 96 120 96 1 1 1 1]
intent_p1 : 0.0
intent_p2 : 0.0
intent_p3 : 0.0
intent_code : none
datatype : int16
bitpix : 16
slice_start : 0
pixdim : [1. 0.19999999 0.19999999 0.19999999 0. 0.
0. 0. ]
vox_offset : 0.0
scl_slope : nan
scl_inter : nan
slice_end : 0
slice_code : unknown
xyzt_units : 10
cal_max : 0.0
cal_min : 0.0
slice_duration : 0.0
toffset : 0.0
glmax : 0
glmin : 0
descrip : b'Rat MRI MRI T2w'
aux_file : b''
qform_code : aligned
sform_code : aligned
quatern_b : 0.0
quatern_c : 0.0
quatern_d : 0.0
qoffset_x : -9.4
qoffset_y : -16.8
qoffset_z : -14.0
srow_x : [ 0.19999999 0. 0. -9.4 ]
srow_y : [ 0. 0.19999999 0. -16.8 ]
srow_z : [ 0. 0. 0.19999999 -13.999999 ]
intent_name : b''
magic : b'n+1'次に、numpyの配列として使いたい場合は get_fdataメソッドを使って型を変換します。
# 読み込んだ画像をNiBabel形式からNumpy.Array形式に変換する
ImgArryData = niimg.get_fdata()こうすることで複数画像同士の演算処理を行うことができるようになります。
画像間の差分をとったり、特定の値で標準化するなど、様々な処理ができるようになりますので、色々と試してみてください。
そしたら最後に画像の書き出し(保存)の方法です。
numpyの配列に変換したものに対して何かしらの編集を行ったというていで話を進めます。
NiBabelでのNIfTIファイルの書き出し(保存)方法

書き出し(保存)の際にはいくつか注意しなければならないことがあります。
ただ書き出す(保存)だけなら簡単です。
しかし、それだとヘッダー情報が空っぽの画像ファイルが生成されてしまいます。
そういったことがないようにヘッダー情報とアフィン変換行列情報を正しく設定した画像の書き出し(保存)が必要です。
まずは元画像のヘッダー情報をコピーしてnew_header変数に入れておきます。
現時点で画像はnumpyの配列になっていますので、これをNIfTIに戻してやる必要があります。
NIfTIに戻すには Nifti1Imageメソッドを使います。
Nifti1Imageメソッドでは第1引数にnumpyの配列、第2引数にアフィン変換行列、第3引数にヘッダー情報を与えます。
アフィン変換行列は.header.get_best_affine()で取得できます。
# 読み込みデータからヘッダーのコピーを作製
new_header = niimg.header.copy()
# NumpyArrayからNIfTI形式に変換する
ConvertMaskImg = nib.Nifti1Image(ImgArryData,
affine=niimg.header.get_best_affine(),
header=new_header)これでConvertMaskImgは元の画像のヘッダーとアフィン変換行列の情報を持ったNIfTIに戻りました。
やりたい処理によってアフィン変換行列は書き換えたりしますが、特に何もなければ上記のように書いてやればOKです。
このひと手間をやっておかないと他のビューワーで表示したとき、正しく表示されませんので注意してください。
ここまで来たらあとは書き出し(保存)だけです。
saveメソッドを使って以下のように記述します。
outputPathNameの部分は出力ファイル名になりますので、適宜変更してください。
出力ファイル名には .nii を付けるのを忘れないでください。
# .niiとして出力
nib.save(ConvertMaskImg, "outputPathName.nii")出力した画像ファイルは必ず他のビューワーで表示を確認するようにしてください。
もしも、自分の意図と異なる表示の場合はソースコードを見直します。
ここまでのパッケージの読み込みから画像の保存までの一連のコードをまとめると以下のようになります。
# パッケージの読み込み
import numpy as np
import nibabel as nib
# load NIfTI image
niimg = nib.load("Schwarz_T2w.nii")
# 読み込んだ画像をNiBabel形式からNumpy.Array形式に変換する
ImgArryData = niimg.get_fdata()
# 読み込みデータからヘッダーのコピーを作製
new_header = niimg.header.copy()
# NumpyArrayからNIfTI形式に変換する
ConvertMaskImg = nib.Nifti1Image(ImgArryData,
affine=niimg.header.get_best_affine(),
header=new_header)
# .niiとして出力
nib.save(ConvertMaskImg, "outputPathName.nii")まとめ
PythonでMRI/CT/PETなどの医用画像を扱うために、NiBabelパッケージの導入方法および画像の読み込み・書き出し(保存)方法について解説しました。
それほど難しい操作はありませんが、NiBabelの使い方について日本語のソースはあまり見かけません。
今回は基本的な使い方のみの紹介でしたが、今後はもう少し突っ込んだ使い方を紹介したいと思います。
Pythonのプログラム・GUIアプリケーションを作って仕事の効率化をお手伝いします!
科研費等での支払いにも対応しており、国立・私立大学の先生から複数の受注実績があります。
対応可能かどうかも含めてお気軽に問い合わせページからご相談ください。
OguLInks公式サイトのトップページに簡単な事例紹介もありますのでご参照ください。

Twitterでお得情報をゲットしたい方はフォローいただけると幸いです。
この記事が役に立ったという方、少額の御寄附をいただけますと小倉のモチベーションアップにつながります。
よろしければ以下より寄付いただけますと幸いですm(_ _)m
すでに購入済みの方はこちら



